Maßarbeit mit Molekülen
Spitze eines Rasterkraftmikroskops manipuliert gezielt molekulare Nanostrukturen.
Physiker und Chemiker der Justus-Liebig-Universität Gießen (JLU) haben eine Methode entwickelt, mit der sie organische Nanoarchitekturen Molekül für Molekül zusammenbauen können. Für die präzise Ausrichtung der Moleküle in der Ebene nutzen sie eine Salzoberfläche, auf der sich die Moleküle relativ leicht bewegen lassen. Als zweites Werkzeug dient die scharfe Spitze eines Rasterkraftmikroskops, die in einem einzelnen Atom oder Molekül endet. Mit diesem „Finger“ werden sukzessive drei Reaktionsschritte durchgeführt: Zunächst werden die molekularen Ausgangsstoffe aktiviert, damit eine spätere intermolekulare Reaktion möglich wird. Im Anschluss werden die zuvor aktivierten molekularen Bausteine auf der Oberfläche zusammengeschoben, bevor die Rastersondenspitze die chemische Reaktion herbeiführt, die zur Bindung der Moleküle führt. Diese Reaktionsschritte werden jeweils mit Hilfe von kurzen Spannungspulsen induziert, welche zwischen der Spitze und der Oberfläche angelegt werden.
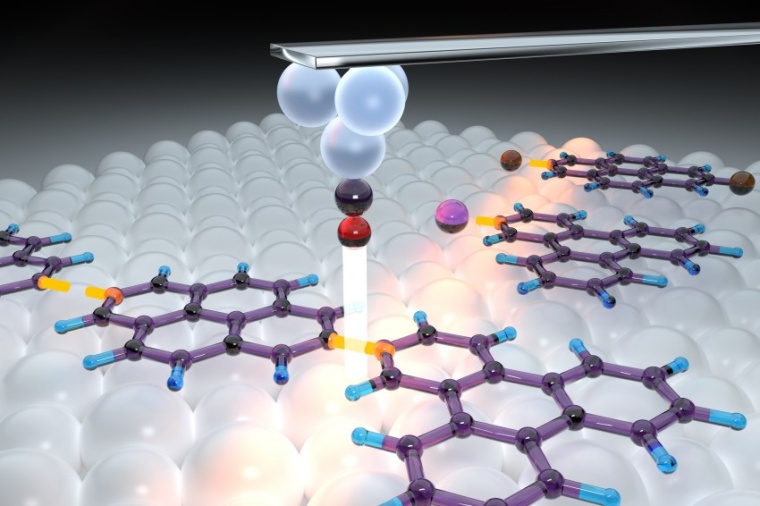
Den Forschern, die im Loewe-Schwerpunkt PriOSS (Prinzipien oberflächengestützter Synthesestrategien) zusammenarbeiten, ist damit die Antwort auf eine Frage gelungen, die Richard Feynman bereits im Jahr 1959 aufwarf und mit der er das Feld der Nanotechnologie begründete: Welche Auswirkungen hätte es, wenn wir neue Materialien Atom für Atom konstruieren könnten? In den frühen 1990er Jahren formulierte der Ingenieur K. Eric Drexler die weitaus kühnere Vision, diese Aufgabe in Zukunft sogar von winzigen Maschinen erledigen zu lassen. Damit wären solche Maschinen in der Lage, nahezu beliebige Nanoarchitekturen – die chemische Stabilität vorausgesetzt – herzustellen und diese mit maßgeschneiderten Eigenschaften auszustatten.
Derartige Vorschläge werden seit Jahrzehnten heftig debattiert. Richard Smalley, Nobelpreisträger für Chemie von 1996, äußerte im Wesentlichen zwei Gegenargumente: Zum einen ließen sich die einzelnen Bausteine (Atome oder Moleküle) mit Hilfe der „Finger“ solcher Fertigungsmaschinen nicht präzise genug ausrichten, da diese Finger selbst nicht unendlich zu verkleinern wären - sie bestünden schließlich auch aus Atomen. Zum anderen würden sowohl die molekularen Bausteine als auch die Produkte aufgrund von Adhäsionskräften stets an den Fingern haften bleiben. Diese triftigen Argumente gingen als das „fat finger“- und das „sticky finger“-Problem in die Wissenschaftsgeschichte ein.
Mit der neuen Methode werden beide Probleme umgangen: Die inerte, das heißt wenig reaktionsfreudige Salzoberfläche übernimmt die Aufgabe einer „non-sticky hand“, und die scharfe Rastersondenspitze ist der „non-fat finger“. So können die JLU-Wissenschaftler in Zukunft neue organische Nanomaterialen herstellen und systematisch untersuchen, wie sich die Struktur auf deren Eigenschaften auswirkt. Damit soll es möglich werden, die Eigenschaften der Nanoarchitekturen gezielt zu beeinflussen. Dies ist besonders interessant für die Anwendung in elektronischen Bauelementen wie organischen Feldeffekttransistoren (OFET), Leuchtdioden (OLEDs, etwa für Smartphonedisplays) oder Solarzellen. Außerdem können durch die schrittweise herbeigeführten chemischen Reaktionen neue Erkenntnisse über die Reaktionsmechanismen von Molekülen auf Oberflächen erlangt werden.
U. Gießen / DE













