3D-Modell für Zelldynamik
Algorithmus sagt das Verhalten lebender Materialien in Raum und Zeit vorher.
Biologische Materialien sind aus einzelnen Komponenten zusammengesetzt, darunter winzige Motoren, die Kraftstoff in Bewegung umwandeln. Auf diese Weise entstehen Bewegungsmuster, und das Material strukturiert sich durch ständigen Energieverbrauch selbst. Die Mechanik von Zellen und Geweben lässt sich mit der Theorie der aktiven Materie beschreiben. Forschende des Max-Planck-Instituts für molekulare Zellbiologie und Genetik in Dresden, des Zentrums für Systembiologie Dresden und der TU Dresden haben nun einen Algorithmus entwickelt und in einem Open-Source Supercomputer-Code implementiert, der die Gleichungen der Theorie aktiver Materie zum ersten Mal in realistischen Szenarien lösen kann. Diese Ergebnisse könnten zeigen, wie Zellen und Gewebe ihre Form erhalten und wie sich künstliche biologische Maschinen designen ließen.
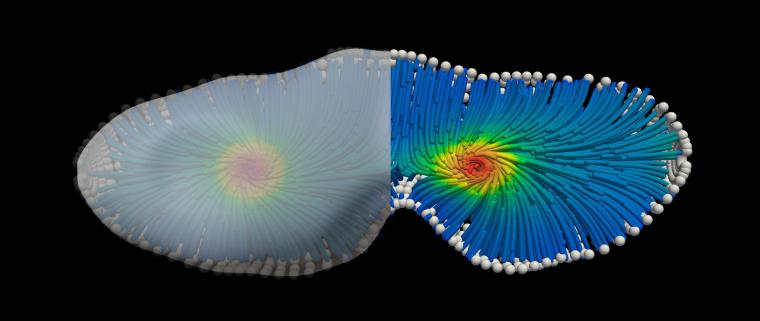
Biologische Prozesse und Muster sind sehr komplex. Physikalische Theorien bieten einen präzisen und quantitativen Rahmen, um diese zu verstehen. Die Theorie der aktiven Materie bietet einen Rahmen, um das Verhalten aktiver Materie zu verstehen und zu beschreiben. Doch erfordern die mathematischen Gleichungen die Leistung von Supercomputern. Es gibt verschiedene Möglichkeiten, das Verhalten aktiver Materie vorherzusagen. Einige konzentrieren sich auf die winzigen einzelnen Teilchen, andere untersuchen aktive Materie auf molekularer Ebene, und wieder andere untersuchen aktive Flüssigkeiten im Großen.
Forschende aus der Arbeitsgruppe von Ivo Sbalzarini haben jetzt einen Computeralgorithmus entwickelt, der die mathematischen Gleichungen aktiver Materie löst. Das Team beschreibt einen Algorithmus, der die komplexen Gleichungen der aktiven Materie in drei Dimensionen und in komplex geformten Geometrien lösen kann. „Unser Algorithmus funktioniert für verschiedene Geometrien in drei Dimensionen und im Zeitverlauf“, sagt Abhinav Singh. „Selbst wenn die Datenpunkte nicht gleichmäßig verteilt sind, nutzt unser Algorithmus einen neuartigen numerischen Ansatz, der nahtlos für komplexe biologisch realistische Szenarien funktioniert, um die Gleichungen der Theorie mit hoher Genauigkeit zu lösen. Mithilfe unseres Ansatzes können wir endlich das langfristige Verhalten aktiver Materialien sowohl in bewegten als auch in unbewegten Szenarien verstehen und ihre Dynamik vorhersagen. Darüber hinaus könnten die Theorie und die Simulationen genutzt werden, um biologische Materialien zu programmieren oder molekulare Maschinen auf der Nanoskala zu entwickeln, die nützliche Arbeit leisten.“
Das Forschungsteam implementierte den Algorithmus mithilfe der Open-Source-Bibliothek OpenFPM. Die Forschenden entwickelten zuerst eine neue Computersprache, die es ermöglicht, Supercomputer-Codes einfacher und viel schneller zu schreiben. Dadurch muss man nicht jedes Mal bei Null anfangen, wenn man einen Code schreiben möchte, wodurch die Entwicklungszeiten in der wissenschaftlichen Forschung effektiv von Monaten oder Jahren auf Tage oder Wochen verkürzt werden können. Aufgrund des großen Rechenaufwands zur Untersuchung dreidimensionaler aktiver Materialien ist der neue Code auf Multiprozessor-Supercomputern skalierbar. Obwohl der Code für leistungsfähige Supercomputern ausgelegt ist, kann er auch auf normalen Arbeitsplatzrechnern für die Untersuchung zweidimensionaler Materialien genutzt werden.
„Zehn Jahre unserer Forschung sind in die Entwicklung dieses Codes geflossen, um die Produktivität computerbasierter Wissenschaft zu verbessern. Dies ist nun alles in einem Werkzeug vereint, um das dreidimensionale Verhalten lebender Materialien zu verstehen. Open-Source, skalierbar und in der Lage, komplexe Szenarien zu bewältigen, eröffnet unser Code neue Möglichkeiten, aktive Materialien zu erforschen. Damit könnten wir endlich verstehen, wie Zellen und Gewebe ihre Form erhalten und damit die grundlegende Frage der Morphogenese beantworten, die Wissenschaftlerinnen und Wissenschaftler seit Jahrhunderten fasziniert. Der Code könnte uns auch dabei helfen, künstliche biologische Maschinen mit minimaler Anzahl von Komponenten zu entwerfen“, sagt Ivo Sbalzarini.
MPI-CBG / JOL











