Eine neue Methode, um Biomoleküle bei der Arbeit zu beobachten, hat ein Forschungsteam vom MPI für Struktur und Dynamik der Materie, der Uni Hamburg und dem European Molecular Biology Laboratory entwickelt. Die „Liquid Application Method for time-resolved Analyses“, kurz LAMA), macht es bedeutend einfacher, enzymatische Reaktionen auszulösen, da hierzu ein Cocktail aus kleinen Flüssigkeitsmengen und Proteinkristallen angewandt wird. Ab dem Zeitpunkt des Mischens werden die Proteinstrukturen in definierten Abständen bestimmt. Mit der dadurch entstehenden Zeitraffersequenz können nun die Bewegungen der biologischen Moleküle abgebildet werden.
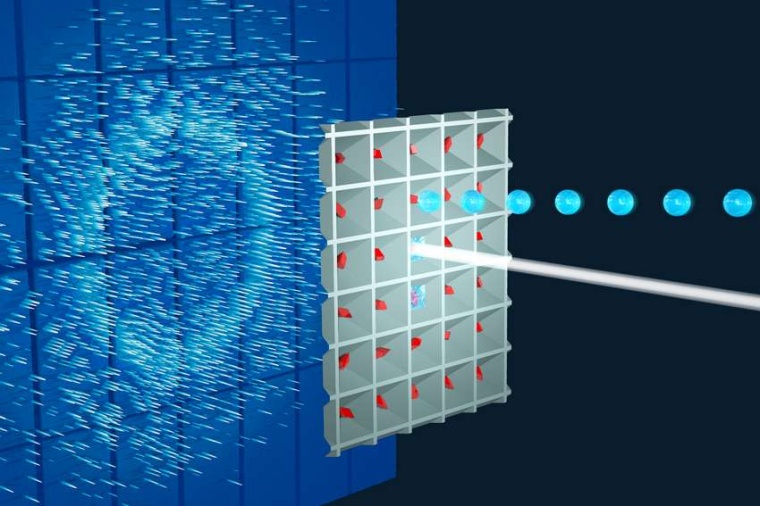
Die Funktionen von Biomolekülen werden nicht nur durch ihre molekularen Strukturen, sondern auch durch deren Veränderungen bestimmt. Mittels der zeitaufgelösten Röntgenkristallographie lassen sich Schnappschüsse eines reagierenden Biomoleküls aufnehmen, die wiederum unser Verständnis dieser dynamischen Bewegungen vertiefen. Allerdings stellt das Auslösen dieser Reaktionen selbst eine große Herausforderung dar, denn typischerweise müssen die Forscher auf optisch ausgelösten Reaktionen mit speziellen Lasern zurückgreifen.
LAMA umgeht diese optischen Auslöser. Sie ist maßgeschneidert für die Untersuchung biologisch relevanter Reaktionszeitskalen, die von wenigen Millisekunden bis hin zu Sekunden oder sogar Minuten andauern. Für Biologen und pharmazeutische Wissenschaftler sind diese Zeitskalen besonders relevant, denn dort geschehen die strukturellen Änderungen, die eine spezifische biologische Funktion oder die Katalyse eines Medikaments bestimmen.
Mit den hochintensiven, mikrofokussierten Röntgenstrahlen der EMBL-Beamline P14-2 am DESY konnte so ein Enzym, das Zucker umwandelt, auf der Millisekundenskala untersucht werden. Durch die neue Methode ließ sich das gesamte Experiment bedeutend einfacher gestalten als mit herkömmlichen Methoden. Einige Pikoliter Zuckerlösung wurden mit Mikrokristallen des Enzyms gemischt. Daraufhin wurden Schnappschüsse der Reaktion aufgenommen, während das Enzym die ringförmige Zuckerstruktur in eine offene Kette umwandelte.
Die neue Methode birgt besonderes Potenzial für hochbrilliante Synchrotron-Strahlungsquellen, da sie Wissenschaftlern nun eine viel größere Anzahl an zeitaufgelösten kristallographischen Untersuchungen ermöglicht. Am PETRA III-Synchrotron des DESY wird die LAMA-Methode schon jetzt als die Standardoption für die neue zeitaufgelöste, makromolekulare Kristallographie-Messstation der EMBL-Beamline P14-2 eingesetzt. Die Nutzung solcher bahnbrechenden Technologien gewährt Forschern bessere Einblicke in biochemische Prozesse und mögliche Antworten auf drängende Umwelt- und Gesundheitsfragen.
MPSD / RK
Weitere Infos
- Originalveröffentlichung
P. Mehrabi et al.: Liquid application method for time-resolved analyses by serial synchrotron crystallography, Nature Methods, online 16. September 2019; DOI: 10.1038/s41592-019-0553-1 - Dynamik in atomarer Auflösung (R. J. D. Miller), Max-Planck-Institut für Struktur und Dynamik der Materie, Hamburg
- European Molecular Biology Laboratory, Hamburg Unit
- Institut für Nanostruktur- und Festkörperphysik, FB Physik, Universität Hamburg
- Centre for Ultrafast Imaging, Universität Hamburg
- PETRA III-Synchrotron, Deutsches Elektronen Synchrotron, Hamburg













