Intramolekulare Abstände in Biomolekülen optisch mit hoher Genauigkeit gemessen
Minflux-Mikroskopie detektiert Strukturen mit einer Präzision im Bereich von einem Ångström.
Einem Team um die Physiker Steffen Sahl und Stefan Hell vom MPI für multidisziplinäre Naturwissenschaften und vom MPI für medizinische Forschung ist es gelungen, mit einem Lichtmikroskop Abstände innerhalb von Biomolekülen bis hinunter zu einem Nanometer zu messen, und zwar mit Ångström-Präzision. Die mit der Minflux-Mikroskopie erreichte intramolekulare Auflösung ermöglicht es, die räumlichen Abstände von Untereinheiten in Makromolekülen optisch zu erfassen und damit unterschiedliche Konformationen einzelner Proteine im Lichtmikroskop nachzuweisen.
Wie sieht es im Nanokosmos lebender Zellen aus? Die Fluoreszenzmikroskopie ermöglicht es, spezifische Moleküle in Zellen oder Geweben sichtbar zu machen und ist aus der biologischen und medizinischen Forschung nicht mehr wegzudenken. Mithilfe neuer höchstauflösender Nanoskopie-Konzepte wie der Minflux-Mikroskopie lassen sich sogar eng benachbarte Biomoleküle lichtmikroskopisch voneinander trennen. Das Innere von Zellen genau abzubilden ist das eine, aber kann man optische Mikroskopie auch auf die Vermessung einzelner Proteine oder anderer Makromoleküle anwenden?
Wie die Forscher jetzt demonstrierten, geht das. Und zwar erstaunlich gut. In ihrer jüngsten Arbeit zeigte das Team, dass sich mit der Minflux-Methode auch der dreidimensionale Abstand zwischen zwei Fluoreszenzmarkern, die jeweils an einer spezifischen Stelle im Makromolekül angebracht sind, optisch vermessen lässt – und zwar mit Ångström-Präzision.
Abstände zwischen zwei räumlich verankerten Fluoreszenzmolekülen von ein paar Nanometern zu vermessen, war mit Minflux zwar denkbar, aber experimentell nicht so einfach umzusetzen oder nachzuweisen. „Bei Abständen kleiner als fünf bis zehn Nanometer wechselwirken die rund ein Nanometer kleinen Fluoreszenzmoleküle häufig miteinander. Dadurch können sie ihr Fluoreszenzlicht nicht unabhängig voneinander emittieren – die Voraussetzung für eine zuverlässige Abstandsmessung“, erklärt Sahl
„Am Beginn unserer Arbeit stand eine Abschätzung: so klein sind Proteine, und so genau können wir prinzipiell werden“, so der Physiker weiter. „Warum realisieren wir nicht Auflösungen innerhalb eines Biomoleküls?“ Denn Nanometerabstände zwischen zwei Proteinen, oder deren Untereinheiten zu erfassen, war bislang der Förster-Resonanzenergietransfer-Methode, kurz FRET, vorbehalten, einer Standardmethode in der Struktur- und Molekularbiologie.
In diesen Auflösungsbereich ist das Team nun auch mit der Minflux-Mikroskopie vorgestoßen. Zum Einsatz kamen eigens dafür am MPI für multidisziplinäre Naturwissenschaften entwickelte photoaktivierbare Fluoreszenzmoleküle, die mit einer geringen Dosis UV-Licht nacheinander angeschaltet werden können, aber nicht miteinander wechselwirken. So ließen sich die zu vermessenden Positionen im Makromolekül mit einem einzelnen Fluoreszenzmolekül markieren und unabhängig voneinander auf Ångström genau erfassen.
„Wir haben gezeigt, dass mit Minflux alle Abstände – bis hin zum unmittelbaren Kontakt der Fluoreszenzmoleküle – gemessen werden können. Dafür reicht es, die direkte Position der Moleküle in zwei oder drei Dimensionen zu bestimmen“, so Sahl. „Mit unseren Experimenten erreichen wir damit den Abstandsbereich von FRET und gehen sogar darüber hinaus.“
FRET dagegen schätzt den Abstand zwischen zwei Farbstoffmolekülen indirekt über den Energieübertrag von einem Farbstoff zum anderen ab. Dabei beeinflussen nicht nur der Abstand, sondern auch die Orientierung der Farbstoffmoleküle das Messergebnis. Das kann zu Unsicherheiten führen, wenn es darum geht, den intramolekularen Abstand präzise zu vermessen. Auch ist das FRET-Verfahren in Studien von Proteinuntereinheiten bislang häufig limitiert, wenn sich diese aus dem messbaren Abstandsbereich herausbewegen. „Hier kann die Minflux-Methode ihre Stärke ausspielen, indem sie alle denkbaren Abstände lückenlos bis hinunter zu einem Nanometer korrekt darstellt“, erklärt Hell. „Minflux ist somit ein neues, sehr leistungsfähiges Werkzeug im Repertoire der Strukturbiologie, um Proteine und andere Biomoleküle und ihre Interaktionen zu untersuchen.“
Für den Nachweis der präzisen Abstandsmessung und Ortsgenauigkeit nutzte das Forschungsteam ein Molekül, mit dem auch die FRET-Methode in einem klassischen Experiment in den 1960er-Jahren ihren Anfang nahm. Damals gelang es Lubert Stryer und Richard Haugland, die von Theodor Förster 1948 publizierte Abstands-Abhängigkeit zu bestätigen. Dazu nahmen sie Polyproline, molekulare Zollstöcke von definierter mittlerer Länge, zu Hilfe. Genau diese Zollstöcke nutzten nun auch die MPI-Forscher in ihren Experimenten – und zeigten, dass sich die Methode prinzipiell sogar in Zellen anwenden lässt.
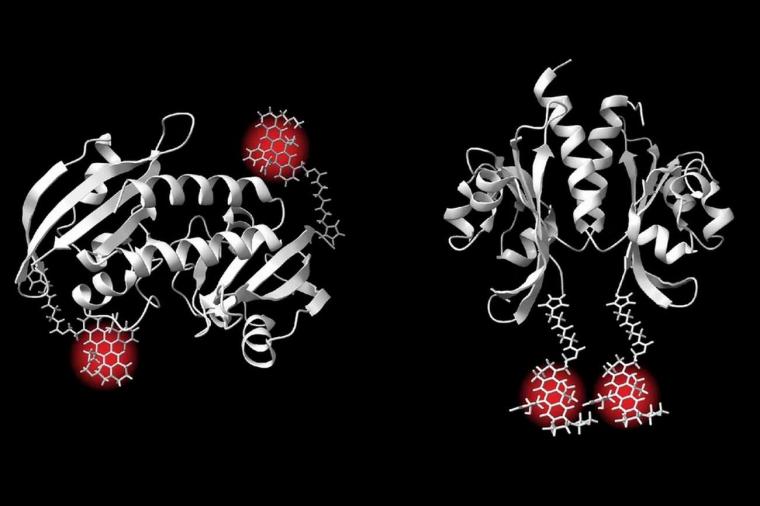
So ließen sich in menschlichen Zellen fluoreszenzmarkierte Lamin-Proteine, die etwa drei Nanometer dünne Filamente an der Membran um den Zellkern ausbilden, mit der Minflux-Methode getrennt abbilden. Darüber hinaus demonstrierten die Wissenschaftler das Potenzial von Minflux anhand von präzisen Abstandsmessungen in weiteren kleinen Proteinen, den Nanobodies, und aus ihnen gebildeten Oligomeren. Am Beispiel von Antikörpermolekülen zeigten die Wissenschaftler, wie mehrere Positionsmessungen es erlauben, die räumliche Lage der Proteinuntereinheiten zueinander aufzulösen.
Mit zwei identischen Untereinheiten des bakteriellen Zitratsensors demonstrierte das Team, dass sogar Abstände von einem Nanometer gemessen werden können. Die Minflux-Mikroskopie detektierte auch die beiden strukturellen Anordnungen der Untereinheiten eindeutig und mit einer Präzision im Bereich von einem Ångström.
MPINAT / RK













