KI schaut in die Zelle
Algorithmus liefert eine quantitative Analyse von zellulären Organellen.
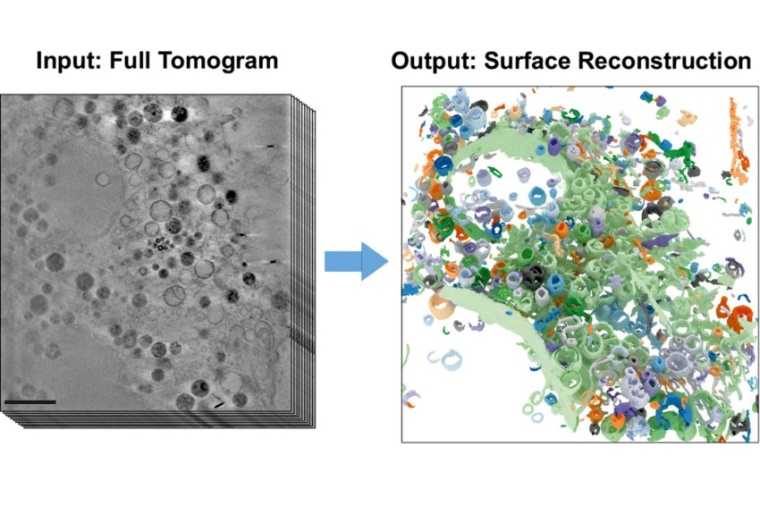
Die Röntgenmikroskopie (Kryo-SXT) ermöglicht hochaufgelöste Einblicke in das Innere von Zellen und Zellorganellen – und das in drei Dimensionen. Bisher wurden die 3D-Datensätze zeitaufwändig manuell analysiert. Nun hat ein Team der Freien Universität Berlin (FU Berlin) einen Algorithmus entwickelt, der auf einem gefalteten neuronalen Netz basiert. Mit Experten aus der Zellbiologie (FU Berlin) und der Röntgenmikroskopie am Helmholtz Zentrum Berlin (HZB) wurde dieser Algorithmus nun erstmals zur Analyse von Zellbestandteilen in Kryo-SXT-Datensätzen eingesetzt und konnte innerhalb weniger Minuten Zellorganellen identifizieren und detailstarke, komplexe 3D-Abbildungen produzieren.
Mit dem hochbrillanten Röntgenlicht von BESSY II lassen sich Mikroskopie-Aufnahmen mit räumlicher Auflösung bis hin zu einigen zehn Nanometern erstellen. Dabei können ganze Zellvolumina untersucht werden, ohne dass eine aufwendige Probenvorbereitung wie bei der Elektronenmikroskopie nötig wäre. Unter dem Röntgenmikroskop erscheinen die winzigen Zellorganellen mit ihren feinen Strukturen und Grenzmembranen deutlich und detailliert, sogar in drei Dimensionen. Daher eignet sich die Kryo-Röntgentomografie hervorragend, um Veränderungen in den Zellstrukturen zu untersuchen, die zum Beispiel durch externe Auslöser verursacht werden.
Die Auswertung der 3D-Tomogramme erforderte jedoch bisher eine weitestgehend manuelle und arbeitsintensive Datenanalyse. Um dieses Problem zu überwinden, haben nun Teams um den Informatiker Frank Noé und den Zellbiologien Helge Ewers (beide FU Berlin) mit der Abteilung Röntgenmikroskopie am HZB zusammengearbeitet. Dabei entwickelte das Informatik-Team einen neuartigen, selbstlernenden Algorithmus. Diese KI-basierte Analysemethode basiert auf der automatisierten Erkennung subzellularer Strukturen und beschleunigt die quantitative Analyse der 3D-Röntgendatensätze. Die Aufnahmen der 3D-Bilder zur Untersuchung des Inneren von biologischen Proben wurden an der U41-Beamline an BESSY II durchgeführt.
„Wir haben in dieser Studie nun gezeigt, wie gut die KI-gestützte Analyse von Zellvolumina funktioniert, und zwar an Säugetierzellen aus Zellkulturen, die so genannte Filopodien besitzen“, sagt Stephan Werner, Experte für Röntgenmikroskopie am HZB. Säugetierzellen besitzen einen komplexen Aufbau mit vielen unterschiedlichen Zellorganellen, die jeweils andere zelluläre Funktionen erfüllen müssen. Filopodien sind dabei Ausstülpungen der Zellmembran und dienen insbesondere der Zellmigration. „Für die Kryo-Röntgenmikroskopie werden die Zellproben zunächst schockgefrostet, und zwar so rasch, dass sich im Inneren der Zelle keine Eiskristalle bilden. Dadurch sind die Zellen in einem nahezu natürlichen Zustand und wir können den strukturellen Einfluss externer Faktoren im Zellinneren studieren“, erklärt Werner.
„Unsere Arbeit hat bereits erhebliches Interesse in der Fachwelt geweckt“, sagt Erstautor Michael Dyhr von der Freien Universität Berlin. Das neuronale Netz erkennt innerhalb kürzester Zeit etwa siebzig Prozent der vorhandenen Zellmerkmale korrekt und ermöglicht damit eine sehr rasche Bewertung des Datensatzes. „Perspektivisch könnten wir mit dieser neuen Analysemethode viel schneller und zuverlässiger als bisher untersuchen, wie Zellen auf Umwelteinflüsse wie zum Beispiel Nanopartikel, Viren oder Karzinogene reagieren“, meint Dyhr.
HZB / DE













