Lichtmikroskop macht Struktur von Hirnneuronen sichtbar
Neue Methode kombiniert Lichtmikroskopie mit dem Einsatz von Hydrogelen und Deep Learning.
Mit Lichtmikroskopen können Forschernde komplizierteste biologischen Strukturen beleuchten. Die komplexen Details und die Architektur des Gehirns zu enträtseln, ist jedoch nach wie vor eine scheinbar unmögliche Aufgabe – handelt es sich doch um Milliarden dicht gepackter Neuronen, von denen jede über jeweils Tausende von Synapsen mit anderen Zellen verbunden ist. Eine neue Mikroskopie-Pipeline namens „LICONN“ (light-microscopy-based connectomics), entwickelt am Institute of Science and Technology Austria (ISTA), bietet nun einen Durchbruch. LICONN ist die erste Technologie, jenseits der Elektronenmikroskopie, die in der Lage ist, Gehirngewebe mit allen synaptischen Verbindungen zwischen Neuronen zu rekonstruieren. Mit ihr lassen sich neben der Struktur der Neuronen auch komplexe molekulare Mechanismen sichtbar machen – und das alles unter Verwendung von Standard-Lichtmikroskopen.
Diese neue Technik wurde von Mojtaba R. Tavakoli, Julia Lyudchik, Johann Danzl und ihren Kolleginnen und Kollegen aus der Danzl Forschungsgruppe entwickelt. Sie arbeiteten dafür mit der Novarino-Gruppe, sowie mit Michal Januszewski und Viren Jain von Google Research zusammen. LICONN funktioniert wie ein akribischer Puzzler, der die komplexen Netzwerke des Gehirns zusammensetzt, indem er die feinsten neuronalen Prozesse zusammenfügt und jede synaptische Verbindung korrekt mit dem entsprechenden Neuron verknüpft. „Bislang war das mit keiner Lichtmikroskopietechnik möglich“, sagt Johann Danzl. „Es war ein langjähriges Ziel unserer Gruppe, eine solche Pipeline für die Rekonstruktion von Hirngewebe zu entwickeln. Und LICONN schafft das, während es bestimmte Moleküle in den Kontext der strukturellen Rekonstruktion einordnet.“
Das Besondere daran ist, dass die Bildaufnahme mit einem handelsüblichen Standardmikroskop erfolgt, das sehr schnell ist und Mehrfarbenaufnahmen ermöglicht. Die Technik kann überall auf der Welt reproduziert werden, da die Forschenden keine hochmodernen, teuren Geräte benötigen, wie sie für aktuelle Ansätze zur Rekonstruktion von Hirngewebe erforderlich wären. Um diesen Detailgrad zu erreichen, muss die Auflösung außerordentlich hoch sein, etwa im Bereich von nur einigen zehn Nanometern. Für LICONN nutzte das Team die chemischen und physikalischen Eigenschaften von Hydrogel, einem dreidimensionalen Polymernetzwerk. Es kann Wasser aufnehmen und so aufquellen, tut dies jedoch auf sehr kontrollierte Weise.
Das zu untersuchende Hirngewebe wird in dieses Hydrogel eingebettet. „Die Zellbestandteile verbinden sich mit dem Hydrogel, sodass die feine Ultrastruktur der Zellen auf das Gel übertragen und für die Mikroskopie erhalten bleibt“, erklärt Danzl. Vor der Bildgebung werden die Strukturen erweitert, indem dem Material Wasser zugeführt wird. Dadurch dehnt sich das Gel in alle Richtungen aus, wobei die relative Anordnung der Gewebestrukturen mit extrem hoher Genauigkeit erhalten bleibt. Zum Vergleich: Herkömmliche Lichtmikroskope sind in ihrer Auflösung klassischerweise auf etwa 250 bis 300 Nanometer begrenzt. Das reicht nicht aus, um dicht gepacktes Hirngewebe zu rekonstruieren. „Durch die Expansion des Hydrogels werden die Strukturen des Hirngewebes so weit auseinandergezogen, dass wir sie mit einem Standard-Lichtmikroskop auflösen können. Diese Methode erhöht die effektive Auflösung um das 16-fache und erreicht eine Auflösung von besser als zwanzig Nanometern“, erklärt Tavakoli.
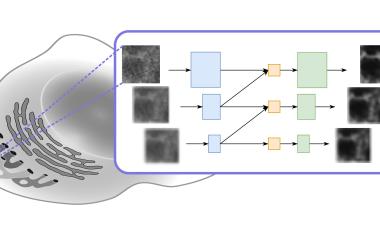
Auch Methoden aus der Informatik spielten eine entscheidende Rolle bei der Entwicklung der Pipeline. Denn die Aufnahme mikroskopischer Bilder führt zur Erfassung zahlreicher Datenpunkte. Die Komplexität der Datensätze spiegelt somit die Komplexität des Gehirns wider. Daher wäre es viel zu aufwendig, alle neuronalen Strukturen in großem Maßstab manuell zu interpretieren und zu rekonstruieren. Deshalb wurden Deep-Learning-Tools von Google Research darauf trainiert, die einzelnen Zellen im Gewebe zu segmentieren. „Durch die Automatisierung der Identifizierung von Neuronen und ihrer komplexen Strukturen in größerem Maßstab mithilfe künstlicher Intelligenz wurde die gewaltige Aufgabe, alle zellulären Komponenten zu rekonstruieren, praktisch beherrschbar“, erklärt Viren Jain von Google Research. „Die Möglichkeit, gleichzeitig bestimmte Moleküle zu visualisieren, liefert eine neue Dimension an Informationen.“
Julia Lyudchik, Doktorandin und Computerwissenschafterin in der Danzl-Gruppe, spielte eine entscheidende Rolle bei der Interpretation der komplexen Datensätze. „Dank der außergewöhnlich hohen Auflösung der Daten war es möglich, die synaptischen Verbindungen zwischen Neuronen automatisch zu erkennen und die Roh-Bilddaten des Gehirns in detaillierte Konnektivitäts-Karten umzuwandeln. Das ist eine komplexe Herausforderung für die Bildverarbeitung“, erklärt Lyudchik. „Außerdem mussten die Methoden sowohl effizient als auch skalierbar sein, da selbst ein kleines Stück Hirngewebe Zehntausende von synaptischen Verbindungen enthalten kann.“
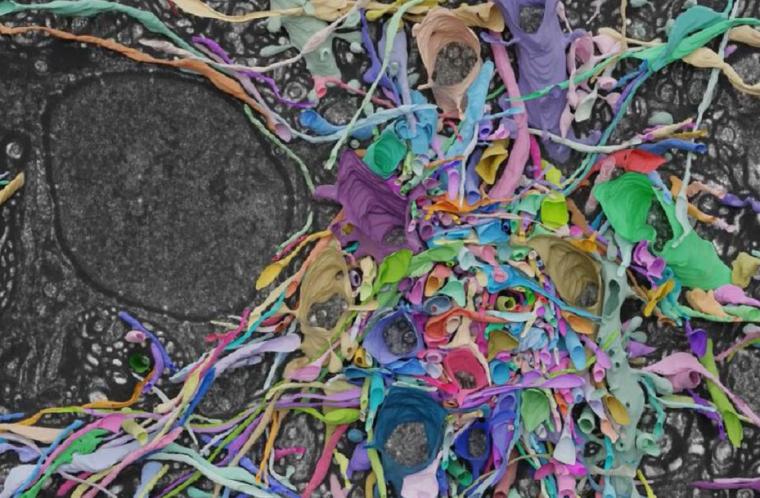
LICONN ermöglicht es, die Position bestimmter Moleküle auf den neuronalen Rekonstruktionen abzubilden, beispielsweise solche, die an der Signalübertragung zwischen Neuronen an Synapsen beteiligt sind. Da Visualisierungen leistungsstarke Werkzeuge sind, um komplexe wissenschaftliche Daten zugänglicher und interpretierbarer zu machen, nutze Lyudchik auch ihre künstlerische Ader dabei, atemberaubende 3D-Darstellungen des Gehirnnetzwerks zu erstellen.
Mithilfe dieses umfassenden Verfahrens können die Forschenden Gehirngewebe akribisch rekonstruieren und neuronale Verbindungen und Netzwerke visualisieren. Das Zusammenspiel zwischen Experimenten und Analysen verschiedener Disziplinen – von der Bildgebung und den Experimenten über die Anwendung fortschrittlicher Deep-Learning-Technologien bei Google Research, bis hin zur computergestützten Analyse – führt zu 3D-Visualisierungen der Gehirnarchitektur auf einem neuen Komplexitätsniveau. „LICONN bringt uns einen Schritt näher daran, die Puzzleteile des Gehirns von Säugetieren zusammenzusetzen und seine Funktionsweise sowohl im gesunden als auch im kranken Zustand besser zu verstehen“, sagt Danzl.
ISTA / JOL