Computermodell optimiert Mikroskopbilder
Neue Deep-Learning-Architektur steigert Kontrast und Auflösung.
Die Lichtmikroskopie offenbart kleinste Details erst mit Hilfe der computergestützten Bildverarbeitung. Obwohl bereits enorme Fortschritte erzielt wurden, gibt es bei der digitalen Verarbeitung weiterhin Entwicklungspotenzial, etwa bei der Erhöhung von Bildkontrast und Auflösung. Ein neues Rechenmodell, das von Forschern des Center for Advanced Systems Understanding (CASUS) am Helmholtz-Zentrum Dresden-Rossendorf und des Max-Delbrück-Centrums für Molekulare Medizin auf der Grundlage einer spezifischen Deep-Learning-Architektur entwickelt wurde, ist schneller als herkömmliche Modelle und erreicht dabei die gleiche oder sogar eine bessere Bildqualität. Das Modell nennt sich Multi-Stage Residual-BCR Net (m-rBCR) und wurde speziell für Mikroskopieaufnahmen entwickelt.
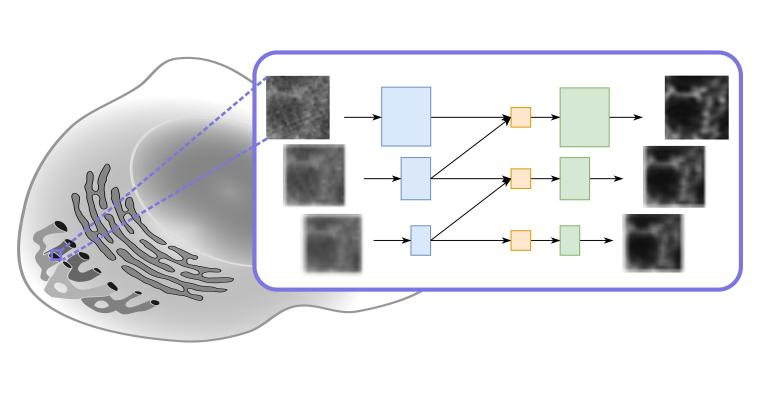
Das neue Modell verleiht der Bildverarbeitungstechnik der Dekonvolution einen ganz neuen Dreh. Dieses rechenintensive Verfahren verbessert Kontrast und Auflösung von digitalen Bildern optischer Mikroskope wie Weitfeld-, Konfokal- oder Durchlichtmikroskope. Die Dekonvolution reduziert die Unschärfe, die durch das jeweils verwendete Mikroskopiesystem verursacht wird. Dafür gibt es hauptsächlich zwei Methoden: die explizite Dekonvolution (direkte Entfaltung) und die auf Deep-Learning-basierende Dekonvolution. Die direkten Entfaltungsansätze basieren auf dem Konzept der Punktspreizfunktion (PSF). Eine PSF beschreibt, wie eine unendlich kleine Lichtquelle, die von der Probe ausgeht, durch das optische System aufgeweitet und in ein dreidimensionales Beugungsmuster verteilt wird. In einem zweidimensionalen Bild befindet sich immer etwas Licht von nicht im Brennpunkt gelegenen Strukturen, die die Unschärfe erzeugen. Kennt man die PSF des mikroskopischen Systems, kann man diese Unschärfe herausrechnen und erhält ein Bild, das der Realität viel näherkommt als das unbearbeitete aufgenommene Bild.
„Das große Problem bei PSF-basierten Entfaltungstechniken ist, dass die PSF eines bestimmten mikroskopischen Systems oft nicht verfügbar oder ungenau ist“, sagt Artur Yakimovich, Leiter einer HZDR-Nachwuchsforschungsgruppe. „Seit Jahrzehnten wird bereits an einer blinden Dekonvolution gearbeitet, bei der die PSF aus dem Bild oder einem Satz von Bildern heraus geschätzt wird. Blinde Dekonvolution ist jedoch nach wie vor ein recht kniffliges Problem und die erzielten Fortschritte sind eher bescheiden.“
Das Yakimovich-Team hat bereits in der Vergangenheit bewiesen, dass das Instrumentarium zur Lösung inverser Probleme im Bereich der Mikroskopie sinnvoll eingesetzt werden kann. Bei inversen Problemen geht es darum, die Ursprungsfaktoren zu ermitteln, die zu bestimmten gemachten Beobachtungen führen. In der Regel sind große Datenmengen und Deep-Learning-Algorithmen erforderlich, um diese Art von Problemen erfolgreich zu lösen. Wie bei den direkten Entfaltungsmethoden erhält man bei der Deep-Learning-basierenden Dekonvolution als Ergebnis Bilder mit höherer Auflösung oder besserer Qualität. Für den auf der ECCV vorgestellten Ansatz verwendeten die Wissenschaftler ein physikbasiertes neuronales Netzwerk namens Multi-Stage Residual-BCR Net (m-rBCR).
Ganz allgemein gibt es für eine Bildverarbeitung zwei Grundvarianten. Sie startet entweder mit der klassischen räumlichen Darstellung eines Bildes oder mit seiner Frequenzversion. Beide Darstellungsformen bieten spezifische Vorteile und je nach Art der Bildbearbeitung nutzt man eher die eine oder die andere Form. Die überwiegende Mehrheit der genutzten Deep-Learning-Architekturen arbeitet mit der räumlichen Darstellung. Sie ist für Fotos gut geeignet. Mikroskopieaufnahmen unterscheiden sich aber davon. Sie sind meist monochromatisch. Bei Techniken wie der Fluoreszenzmikroskopie kommen zum Beispiel spezifische Lichtquellen auf einem schwarzen Hintergrund zum Einsatz. m-rBCR nutzt daher die Frequenzdarstellung als Startpunkt.
„Die Nutzung der Frequenzdomäne kann gerade in solchen Fällen dazu beitragen, optisch aussagekräftige Datenrepräsentationen zu erstellen – ein Konzept, das es m-rBCR ermöglicht, die Entfaltungsaufgabe im Vergleich zu anderen modernen Deep-Learning-Architekturen mit überraschend wenig Parametern zu meistern“, sagt Rui Li. Li schlug vor, die neuronale Netzwerkarchitektur des Modells BCR-Net weiterzuentwickeln. Dieses Modell wurde wiederum von einem frequenzdarstellungsbasierten Signalkomprimierungsschema inspiriert, das in den 1990er Jahren von Gregory Beylkin, Ronald Coifman und Vladimir Rokhlin eingeführt wurde – daher der Name BCR-Transform.
Das Team validierte das m-rBCR-Modell anhand von vier verschiedenen Datensätzen, zwei simulierten Mikroskopie-Bilddatensätzen und zwei realen Mikroskopiedatensätzen. Verglichen mit den neuesten Deep-Learning-basierten Modellen zeigt es eine hohe Leistungsfähigkeit mit deutlich weniger Trainingsparametern und einer kürzeren Laufzeit. Darüber hinaus übertrifft es auch explizite Entfaltungsmethoden deutlich. „Diese neue Architektur nutzt einen bislang wenig beachteten Ansatz, um Darstellungen jenseits der klassischen neuralen Faltungsnetzwerke zu ermöglichen“, sagt Misha Kudryashev, Leiter der Arbeitsgruppe „In-situ-Strukturbiologie“ am Max-Delbrück-Centrum für Molekulare Medizin in Berlin. „Unser Modell reduziert potenziell redundante Parameter erheblich. Wie die Ergebnisse zeigen, geht dies jedoch nicht mit einem Leistungsverlust einher. Das Modell eignet sich explizit für mikroskopische Aufnahmen und stellt aufgrund seiner schlanken Architektur den Trend zu immer größeren Modellen, die entsprechend höhere Rechenleistung erfordern, in Frage.“
Die Yakimovich-Gruppe hat kürzlich ein Modell zur Verbesserung der Bildqualität präsentiert, das auf generativer KI basiert. Ihr Conditional Variational Diffusion Model (CVDM) erzielt Ergebnisse, die dem neuesten Stand der Technik entsprechen und auch das hier vorgestellte m-rBCR-Modell übertreffen. „Allerdings benötigt man dafür Trainingsdaten und entsprechende Rechenressourcen, einschließlich leistungsfähiger Grafikprozessoren, die heutzutage sehr gefragt sind“, sagt Yakimovich. „Das leichtgewichtige m-rBCR-Modell hat diese Einschränkungen nicht und liefert trotzdem sehr gute Ergebnisse. Ich bin daher zuversichtlich, dass wir von der Forschungsgemeinschaft viel Zuspruch erhalten werden. Auch aus diesem Grund haben wir damit begonnen, die Benutzerfreundlichkeit zu optimieren.“
Die Forschenden haben sich zum Ziel gesetzt, das komplexe Netzwerk molekularer Interaktionen zu verstehen, das nach der Infektion mit Krankheitserregern im Körper aktiv ist. Dabei ist die Nutzung der neuen Möglichkeiten des maschinellen Lernens zentral. Zu den Interessensgebieten gehören die Verbesserung der Bildauflösung, die 3D-Bildrekonstruktion, die automatisierte Krankheitsdiagnose und die Bewertung der Rekonstruktionsqualität von Bildern.
HZDR / JOL
Weitere Infos
- Originalveröffentlichung
R. Li et al.: Solving the Inverse Problem of Microscopy Deconvolution with a Residual Beylkin-Coifman-Rokhlin Neural Network, Computer Vision – ECCV 2024, Lecture Notes in Computer Science 15133, 378 (2024); DOI: 10.1007/978-3-031-73226-3_22 - Center for Advanced Systems Understanding (CASUS), Helmholtz-Zentrum Dresden-Rossendorf HZDR, Dresden













