Mikroskopie mit extremer Auflösung
Neuer Ansatz in der Superauflösungsmikroskopie reicht bis zur Angströmskala.
Der Forschungsgruppe um Ralf Jungmann an der Ludwig-Maximilians-Universität München (LMU) und am Max-Planck-Institut für Biochemie (MPIB) ist ein Durchbruch in der Fluoreszenzmikroskopie gelungen. Das Team entwickelte eine Methode namens „Resolution Enhancement by Sequential Imaging“ (RESI), eine revolutionäre Technik, die die Auflösung der Fluoreszenzmikroskopie bis auf die Angströmskala verbessert. Diese Innovation wird einen Paradigmenwechsel bei der Untersuchung biologischer Systeme mit bisher unerreichter Detailgenauigkeit einleiten.
Zellen, die grundlegenden Einheiten des Lebens, enthalten eine Vielzahl komplexer Strukturen, Prozesse und Mechanismen, die lebende Systeme aufrechterhalten und fortbestehen lassen. Viele zelluläre Kernelemente wie DNA, RNA, Proteine und Lipide sind nur wenige Nanometer groß – erheblich kleiner als die Auflösungsgrenze der herkömmlichen Lichtmikroskopie. Die genaue Zusammensetzung und Anordnung dieser Moleküle und Strukturen ist daher oft unbekannt, was zu einem mangelnden mechanistischen Verständnis grundlegender Aspekte der Biologie führt.
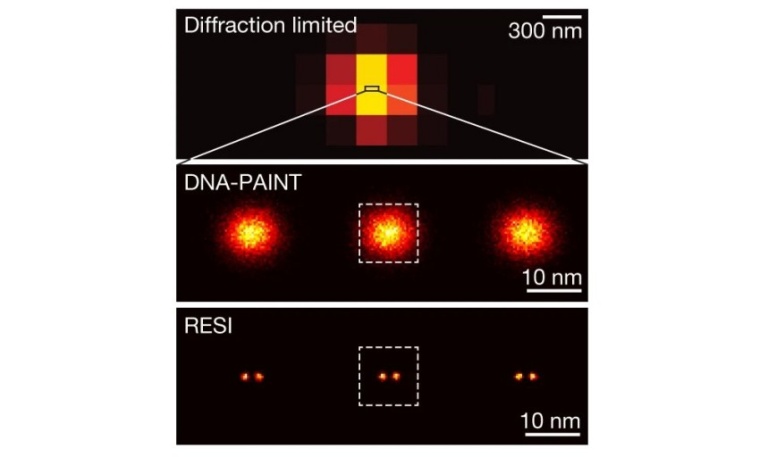
In den letzten Jahren haben Superauflösungstechniken enorme Fortschritte gemacht und erlauben es, subzelluläre Strukturen unterhalb der klassischen Beugungsgrenze des Lichts aufzulösen. Die Einzelmoleküllokalisierungsmikroskopie (Single-Molecule Localization Microscopy, SMLM) ist ein Verfahren, mit dem Strukturen in der Größenordnung von zehn Nanometern aufgelöst werden können, indem ihre individuellen Fluoreszenzemissionen zeitlich getrennt werden. Da einzelne Zielmoleküle in einem sonst dunklen Sichtfeld stochastisch aufleuchten (blinken), können ihre Positionen mit einer Genauigkeit unterhalb der Beugungsgrenze bestimmt werden. DNA-PAINT, erfunden von der Jungmann-Gruppe, ist eine SMLM-Technik, die temporäres Hybridisieren von Farbstoff-markierten DNA-„Imager“-Strängen nutzt, um das notwendige Blinken für Superauflösung zu erreichen. Bislang war jedoch selbst DNA-PAINT nicht in der Lage, die kleinsten zellulären Strukturen aufzulösen.
In der aktuellen Studie, die von den Erstautoren Susanne Reinhardt, Luciano Masullo, Isabelle Baudrexel und Philipp Steen zusammen mit Jungmann geleitet wurde, stellt das Team einen neuen Ansatz in der Superauflösungsmikroskopie vor, der eine grundsätzlich „unbegrenzte“ räumliche Auflösung ermöglicht. Die neue Technik mit der Bezeichnung „Resolution Enhancement by Sequential Imaging“ (RESI) nutzt die Fähigkeit von DNA-PAINT, die Identität von Zielobjekten durch eindeutige DNA-Sequenzen zu kodieren. Nahe aneinanderlegende Moleküle, die mit klassischer SMLM nicht aufgelöst werden können, werden durch unterschiedliche DNA-Sequenzen markiert. Dadurch entsteht ein zusätzliches Unterscheidungsmerkmal.
Durch sequenzielle Bildgebung erst einer und dann der anderen Sequenz (und damit der jeweiligen Moleküle) können sie nun eindeutig getrennt und aufgelöst werden. Da sie nacheinander abgebildet werden, können die Ziele beliebig nahe beieinander liegen, was mit keiner anderen Technik möglich ist. Darüber hinaus erfordert RESI keine spezialisierten Mikroskope, es kann mit jedem Standard-Fluoreszenzmikroskop angewendet werden, was es für fast alle Forscher leicht zugänglich macht.
Um die Auflösungsverbesserung von RESI zu demonstrieren, stellte sich das Team der Herausforderung, einen der kleinsten räumlichen Abstände in einem biologischen System aufzulösen: den Abstand zwischen einzelnen Basen entlang einer DNA-Doppelhelix, die weniger als einen Nanometer voneinander entfernt sind. In einer DNA-Origami-Nanostruktur, die einzelsträngige DNA-Sequenzen in einem Abstand von nur einem Basenpaar enthielt, konnte das Forscherteam einen Abstand von 0,85 Nanometer zwischen benachbarten Basen auflösen. Den Forschern gelang diese Messung mit einer Präzision von einem Angström, was die beispiellosen Möglichkeiten des RESI-Verfahrens unterstreicht.
Wichtig ist, dass die Technik universell und nicht nur auf Anwendungen in DNA-Nanostrukturen beschränkt ist. Um das zu zeigen, untersuchte das Team den molekularen Wirkmechanismus von Rituximab, einem Anti-CD20 monoklonalen Antikörper, der erstmals 1997 zur Behandlung von CD20-positivem Blutkrebs zugelassen wurde. Die Untersuchung der Auswirkungen solcher Medikamentenmoleküle auf molekulare Rezeptormuster übersteigt jedoch die räumliche Auflösung herkömmlicher Mikroskopiertechniken. Zu verstehen, ob und wie sich solche Muster im Krankheitsfall sowie bei einer Behandlung verändern, ist nicht nur für die mechanistische Grundlagenforschung, sondern auch für die Entwicklung neuer zielgerichteter Krankheitstherapien von großer Bedeutung.
Mit RESI konnten Jungmann und sein Team die natürliche Anordnung von CD20-Rezeptoren in unbehandelten Zellen als Dimere offenlegen und aufdecken, wie sich CD20 bei Medikamentenbehandlung zu Ketten von Dimeren umorganisierte. Die Erkenntnisse auf der Einzelproteinebene helfen nun, die molekulare Wirkweise von Rituximab besser zu verstehen. Da RESI in ganzen, intakten Zellen durchgeführt wird, schließt die Technik die Lücke zwischen rein strukturellen Methoden wie Röntgenkristallographie oder kryogener Elektronenmikroskopie und herkömmlichen bildgebenden Verfahren mit geringerer Auflösung für ganze Zellen.
LMU / DE
Weitere Infos
- Originalveröffentlichung
S. C. M. Reinhardt et al.: Ångström-resolution fluorescence microscopy, Nature 617, 711 (2023); DOI: 10.1038/s41586-023-05925-9 - Molecular Imaging and Bionanotechnology (R. Jungmann), Ludwig-Maximilians-Universität München











