Molekulare Mechanismen für die Arzneimittel-Entwicklung
Dynamik von Bindungs- und Abdiffusions-Prozessen auf einer Zeitskala von Sekunden bis zu einer halben Minute vorhersagbar.
Molekulardynamik-Simulationen sind in den modernen Biowissenschaften allgegenwärtig geworden. Dabei werden die Wechselwirkungen zwischen Atomen und Molekülen sowie deren sich daraus ergebenden räumlichen Bewegungen schrittweise berechnet und dargestellt. Wissenschaftler versuchen derzeit, bei diesem Analyseprozess den Zugang zu biologisch relevanten Längen- und Zeitskalen zu erlangen. Das ist nötig, um molekulare Prozesse wie Proteinfaltung und Protein-Medikamentenbindung zu beschreiben, was zum Beispiel für die moderne Arzneimittel-Entwicklung entscheidend ist. Einem Team um Steffen Wolf und Gerhard Stock an der Uni Freiburg ist es jetzt gelungen, in pharmakologisch relevanten Testsystemen die Dynamik von Bindungs- und Abdiffusionsprozessen auf einer Zeitskala von Sekunden bis zu einer halben Minute vorherzusagen.
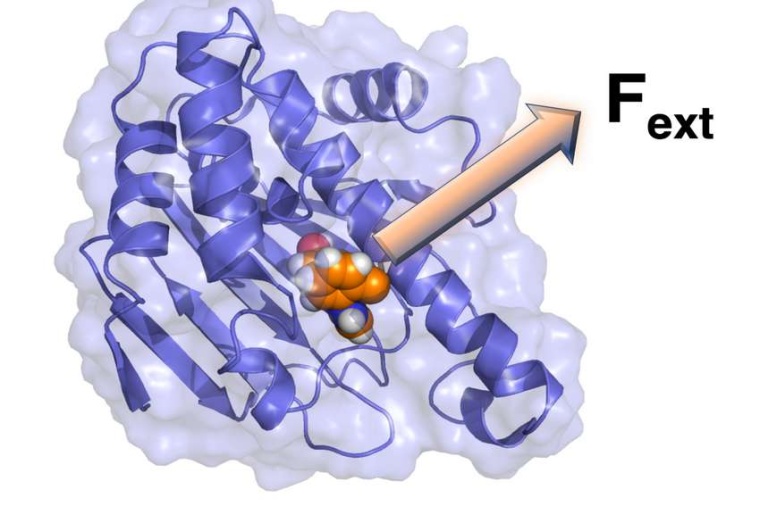
Aufgrund der Notwendigkeit, atomistische Simulationen mit einer zeitlichen Auflösung von Femtosekunden durchzuführen, können Forscher Vorgänge, die Sekunden und mehr benötigen, wie zum Beispiel das Binden und Lösen von Wirkstoffen an und von ihrem jeweiligen Zielprotein, noch nicht explizit berechnen. Ein möglicher Ansatz, um Simulationen zu beschleunigen ist die Grobkörnung der gesamten Systemdynamik, die eine Domäne der statistischen Mechanik des Nichtgleichgewichts ist.
Dafür müssen langsame Prozesse wie die Protein-Liganden-Abdiffusion und schnelle Prozesse wie Protein-Vibrationen oder Wasserfluktuationen zeitlich getrennt werden. Erst dann können die Wissenschaftler die Langevin-Gleichung verwenden, eine stochastische Differentialgleichung, mit der die Dynamik entlang der relevanten langsamen Freiheitsgrade eines physikalischen Systems beschrieben wird. Mit dieser Gleichung stellen sie die Dynamik des Systems entlang einer Reaktionskoordinate wie der Entfernung des Liganden von seiner Bindungsstelle dar. Alle anderen, schnelleren Bewegungen werden als Reibung berücksichtigt.
Um diese erforderliche Vereinfachung der Systemdynamik zu erreichen, haben die Physiker die dissipationskorrigierte „targeted“ Molekulardynamik, kurz dcTMD, entwickelt und mittels Rechnungen auf dem Hochleistungsrechner BinAC an der Uni Tübingen getestet. Durch Anwendung einer Zwangskraft, um ein System aktiv entlang einer interessierenden Koordinate zu ziehen, kann die erforderliche Zugarbeit in freie Energie und Reibungsfelder des Prozesses zerlegt werden. In der aktuellen Studie zeigen die Forscher, dass diese dcTMD-Felder als Input für eine Simulation der Langevin-Gleichung entlang des Zugkoordinators verwendbar sind.
Dadurch konnten sie die notwendige Rechenleistung stark reduzieren. Die Simulationszeit von einer Millisekunde ist so innerhalb von einigen Stunden auf einem einzigen Rechenkern eines Standard-Desktop-Computers erreichbar. Zudem verändern Langevin-Felder im Gegensatz zu vollständig atomistischen Proteinen bei höheren Temperaturen nicht ihre Struktur. „Daher können Hochtemperatursimulationen eine beschleunigte Dynamik erzeugen. Diese können wir nutzen, um die Dynamik bei einer niedrigeren interessierenden Temperatur wiederherzustellen, bei der sich die Felder aus gezielten MD-Simulationen ableiten“, so Stock.
Die Wissenschaftler haben die die Aufspaltung von Natriumchlorid und von zwei Protein-Ligand-Komplexen als Testsysteme verwendet. In diesen gelang es ihnen, die Dynamik von Bindungs- und Entbindungsprozessen auf einer Zeitskala von Sekunden bis zu einer halben Minute vorherzusagen. „Während die Langevin-Felder nur aus Entbindungssimulationen erzeugt wurden, konnten sie sowohl Entbindungs- als auch Bindungskinetiken innerhalb eines Faktors 20 und Dissoziationskonstanten innerhalb eines Faktors 4 vorhersagen, was im Vergleich mit anderen Vorhersagemethoden im besten erreichbaren Bereich liegt“, erläutert Wolf.
Gleichzeitig erfordere dieser neue dcTMD-Ansatz nur ein Zehntel der Rechenleistung als andere Vorhersagemethoden. Nicht zuletzt erlaube die Bestimmung von Reibungsprofilen einen Einblick in molekulare Prozesse, die die freie Energie nicht aufzeigt, betonen die Forscher. In allen untersuchten Systemen scheint die Bildung einer Hydratationsschale aus Wasser die Hauptquelle der Reibung zu sein. Das erlaubt, neue Möglichkeiten für das Design von Medikamenten mit gewünschter Bindungs- und Abdiffusionskinetik abzuleiten.
ALU / RK
Weitere Infos
- Originalveröffentlichung
S. Wolf et al.: Multisecond ligand dissociation dynamics from atomistic simulations, Nat. Comm. 11, 2918 (2020); DOI: 10.1038/s41467-020-16655-1 - Biomolekulare Dynamik, Institut für Physik, Albert-Ludwigs-Universität Freiburg im Breisgau













