Wie gut funktionieren Biomarker?
Neue Methode zeigt, wie zuverlässig sich Zielproteine in der superauflösenden Fluoreszenz-Mikroskopie markieren lassen.
Moderne Mikroskopie-Verfahren erlauben es, das Innenleben von Zellen in erstaunlichem Detailreichtum zu untersuchen. „Inzwischen können wir die Anordnung und Interaktion einzelner Proteine unter dem Mikroskop beobachten“, sagt Ralf Jungmann, Leiter des Lehrstuhls für molekulare Physik des Lebens an der Uni München. Das Team des Biophysikers hat kürzlich die revolutionäre Methode RESI entwickelt, das „Resolution Enhancement by Sequential Imaging“. Damit lässt sich die Auflösung der Fluoreszenz-Mikroskopie bis auf die Ångströmskala verbessern – weit unterhalb der klassischen Beugungsgrenze des Lichts. Ausschlaggebend dafür sind DNA-konjugierte Markermoleküle, welche die Forscher zielgenau an den Molekülen anbringen, die sie besser verstehen wollen.
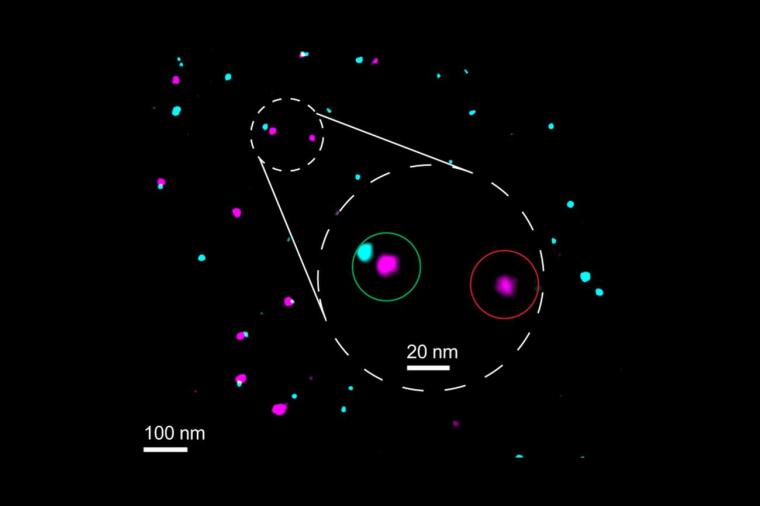
Jetzt hat Jungmanns Team eine Technik vorgestellt, mit der sich quantifizieren lässt, wie gut die Bindung von Biomarkermolekülen an die Zielproteine funktioniert. „Das ist absolut ausschlaggebend, wenn man quantitativ belastbare Aussagen treffen will“, erklärt der Physiker. Kenne man die Markierungseffizienz, so könne man auf diese Weise räumlich aufgelöste Proteomik betreiben. So finde man nicht nur heraus, was einzelne Proteine in einer Zelle machen, sondern auch in welchem Ausmaß sie vorhanden sind und wie sich ihre Menge und ihr Verhalten unter bestimmten Umständen verändern. „Das geht aber eben nur, wenn wir einschätzen können, wie gut die Markierung geklappt hat.“ Denn nur markierte Proteine blinken unter dem Mikroskop auf und werden so sichtbar.
Die von Jungmanns Team entwickelte Methode ermöglicht diese Einschätzung, indem sie die Zielproteine zusätzlich mit einem Referenzbiomarker versieht. Er leuchtet beim Mikroskopieren in einer anderen Farbe, sodass erfolgreich markierte Proteine zweifarbig erscheinen. Jungmanns Team hat das unter anderem anhand des Membranproteins CD86 demonstriert: Die Referenz erzeugt eine rosafarbene Fluoreszenz, der eigentliche Marker eine bläuliche. So entsteht ein Muster unzähliger pinker und blauer Lichtpunkte. Wo die Markierung nicht geklappt hat, leuchtet lediglich die Referenz einzeln auf. Aus dem Verhältnis doppelt und einzeln leuchtender Moleküle errechnet sich die Markierungseffizienz.
Das Verfahren bietet mehrere Vorteile im Vergleich zu bisherigen Methoden zur Bestimmung der Bindeeffizienz. „Es funktioniert nicht nur in vitro, sondern auch in vivo, also im Kontext intakter Zellen“, erklärt Jungmann. „Die Technik ist außerdem auf eine Vielzahl verschiedener Zielmoleküle, Biomarker und Proben anwendbar und kompatibel mit einer ganzen Reihe superauflösender Methoden.“ Eine zuverlässige und breit einsetzbare Möglichkeit zur Einschätzung der Marker-Effizienz sei von entscheidender Bedeutung, um eine präzise Datenauswertung zu gewährleisten und zuverlässige Vergleiche zwischen verschiedenen Bindern, Markierungsbedingungen und Forschungslabors zu ermöglichen.
Die Wissenschaftler sind sich sicher, dass mit der neuen Quantifizierungsmethode nun der Weg geebnet ist, das Potenzial ihrer superauflösenden Mikroskopie deutlich zu erweitern. „Jetzt können wir auch konkrete biomedizinische Anwendungen ins Auge fassen, bei denen eine quantitative Erfassung der Proteine und Prozesse von großer Bedeutung ist“, sagt Jungmann. Dazu gehört zum Beispiel die Krebsforschung, wo Informationen über Interaktionen zwischen Proteinen auf der Zelloberfläche und Medikamenten mit molekularer Auflösung bei der Entwicklung neuartiger Medikamente essenziell sind.
LMU / RK













