Extrem genauer Blick in lebende Zellen
Neue Methode erlaubt räumliche Auflösung von weniger als zehn Nanometer.
Forschende der Universität Würzburg entwickeln mit dem „Photoswitching Fingerabdruck“ eine einzigartige Technologie, die erstmals die Untersuchung molekularer Vorgänge und der Regulation einzelner Proteine in lebenden Zellen mit einer räumlicher Auflösung von weniger als zehn Nanometer ermöglicht. Die Anwendung erstreckt sich von der biologischen bis hin zur medizinischen Forschung.
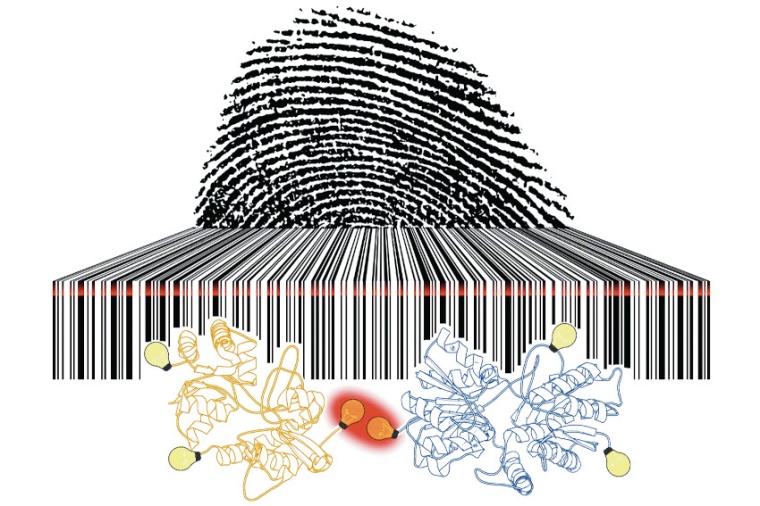
Die hochauflösende Fluoreszenzmikroskopie erlaubt es, Fluoreszenzbilder von Zellen, Organellen und Molekülkomplexen mit bisher unerreichter räumlicher Auflösung aufzunehmen. Diese Auflösung reicht jedoch nicht, um Proteine mit einer Größe von wenigen Nanometern und ihre Wechselwirkungen mit anderen Molekülen oder die Architektur von Proteinkomplexen aufzulösen. Es limitiert zum Beispiel die Erforschung des molekularen Zusammenspiels der Neuronen in Lern- und Gedächtnisprozessen. Entwickelt von der Forschungsgruppe von Markus Sauer und Gerti Beliu, ermöglicht der neue Photoswitching Fingerabdruck die optische Darstellung von dynamischen Wechselwirkungen mit anderen Molekülen in der Zelle. „Bisher existiert keine Methode, die eine strukturelle optische Auflösung in Zellen im sub-10-Nanometer Bereich verlässlich erlaubt. Durch die Aufklärung dieser Barriere zugrundeliegenden Ursache ist es uns erstmals gelungen, in Kombination mit neuen direkten Markierungsmethoden, eine zelluläre Auflösung von wenigen Nanometern zu realisieren. Dieser Fortschritt ermöglicht die Aufklärung molekulare Funktionen und der Architektur wichtiger Komponenten unserer Zellen“, sagt Sauer.
Einzelmolekül-Lokalisations-Mikroskopiemethoden wie dSTORM ermöglichen Auflösungen im Bereich von zehn bis zwanzig Nanometer. In Kombination mit strukturierten Beleuchtungsverfahren konnten Lokalisationsgenauigkeiten von bis zu einem Nanometer für Farbstoffe erreicht werden. Leider konnte diese hohe Lokalisationspräzision nicht in eine räumliche Auflösung von wenigen Nanometern in Zellen übersetzt werden. Das Problem: Die gängigen Markierungsmethoden, zum Beispiel Immunfärbungen mit einem Antikörper, verursachen einen Abstandsfehler von mehr als zehn Nanometer. Dadurch verhindert die Größe der Markierungsmoleküle eine Auflösung im Nanometerbereich.
Die Ursache für die Auflösungsbarriere war bisher nicht bekannt. „Wir konnten nun erstmals zeigen, dass die Photoschaltraten der Farbstoffe zwischen einem An- und Aus-Zustand bei Abständen unterhalb von zehn Nanometer aufgrund verschiedener Energietransferprozesse zwischen Farbstoffen stark beeinflusst wird. Hierdurch kommt es während den ersten Sekunden eines Experiments gehäuft zu An-Zuständen und damit verbunden zum schnellen Photobleichen der Farbstoffe, was ihre individuelle Lokalisation erschwert“, erklärt Sauer. „Die verringerte Lokalisationswahrscheinlichkeit der Farbstoffe resultiert daher in einer schlechteren strukturellen Auflösung, als man aufgrund der individuellen Lokalisationsgenauigkeit erwarten würde. Dies ähnelt einem Orchester, indem alle Instrumente zeitgleich am Anfang des Stücks ihre Beiträge spielen; es ist unmöglich die einzelnen Tonspuren herauszuhören.“
Der Photoswitching Fingerabdruck und die Fluoreszenzabklingdauer enthalten aber auch Information über die Anzahl der vorhandenen Farbstoffe und, aufgrund der Abstandsabhängigkeit der Energietransferprozesse, auch Information über deren Abstände, ohne dass man die einzelnen Farbstoffe optisch auflösen kann. Durch den Einbau unnatürlicher Aminosäuren in multimere Membranrezeptoren durch Erweiterung des genetischen Codes mit anschließender bioorthogonaler Click-Markierung mit kleinen Fluoreszenz-Farbstoffen konnten die Würzburger Forschungsgruppen nun im nächsten Schritt aufzeigen, wie die spezifische ortsgenaue Markierung von Proteinen in Zellen ohne Abstandsfehler mit Sub-10-Nanometer Abständen gelingt. „Durch die Analyse der Photoswitching Fingerprints der multimeren Rezeptoren in der Plasmamembran konnten wir so erstmals Abstände zwischen Rezeptoruntereinheiten im Bereich von fünf bis sieben Nanometer in Zellen abschätzen und die Anzahl der markierten Untereinheiten bestimmen“, sagt Beliu.
Im nächsten Schritt will das Forschungsteam die Photoswitching Fingerprint Analyse optimieren und in Kombination mit der Einzelmolekül-Lokalisations-Mikroskopie mittels strukturierter Beleuchtung und DNA-PAINT zum verlässlichen Super-Resolution Imaging in Zellen mit sub-10-Nanometer Auflösung einsetzen. Dies soll neue Erkenntnisse in der molekularen Organisation zellulärer Strukturen, Organellen und Multiproteinkomplexe sowie der Strukturaufklärung von Proteinkomplexen mit optischen Methoden ermöglichen.
Die neu entwickelte Methode bietet nicht nur einzigartige Einblicke in molekulare Mechanismen der Infektions-, Lipid- und Krebsforschung: Der Photoswitching Fingerabdruck hat auch das Potential, die Dynamik und Komplexität von Rezeptoren im Nervensystem, die an den Synapsen der Neurone für die Signalweiterleitung wichtig sind, realistischer darzustellen. Dieses Zusammenspiel der Neuronen definiert unsere täglichen Lern- und Gedächtnisprozesse. „Es ist daher fundamental wichtig zu verstehen, wie sich dieses molekulare Orchester zusammensetzt und funktioniert“, sagt Beliu.
U. Würzburg / JOL











