Proteinkristalle mit Elektronen abbilden
Schnellere Bestimmung von Proteinstrukturen per serieller Elektronenbeugung.
Um die biologischen Funktionen von Proteinen zu verstehen, ist es unerlässlich, ihre Struktur zu erforschen. Durch ihrer winzigen Dimensionen und Zerbrechlichkeit sind diese Strukturen jedoch schwer zu bestimmen. Die immense Dosis an hochenergetischer Röntgenstrahlung, die Daten in ausreichender Auflösung erzeugt, richtet in den zu untersuchenden Proteinen große Schäden an, die oft die Strukturauflösung verhindert. Jetzt haben Forscher vom MPI für Struktur und Dynamik der Materie und dem DESY eine innovative neue Methode entwickelt, die diese Probleme umgeht und weit verbreitete, kosteneffektive Technologien nutzt.
Seit Jahrzehnten suchen Forscher aus unterschiedlichen Arbeitsgebieten bereits nach kreativen Lösungen für das Strahlungsschäden-
Die Forscher vom MPSD und vom DESY haben auf raffinierte Weise diese Ansätze mit Big Data-
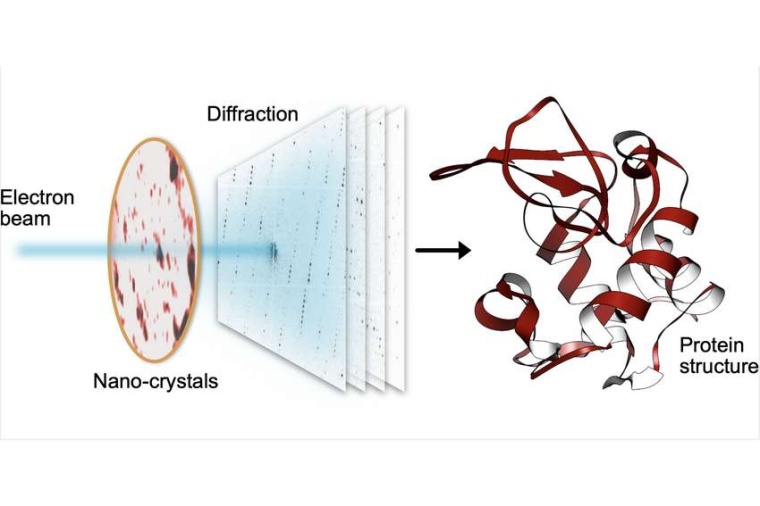
Statt ein Großforschungsinstrument wie EuXFEL einzusetzen, verteilten sie die Kristalle auf einem dünnen Kohlefilm und legten sie in ein Elektronenmikroskop, wie es in vielen Laboren vorhanden ist. Der Elektronenstrahl wird dazu gebracht, von einem Nanokristall zum nächsten zu hüpfen, um Beugungsdaten zu sammeln. Die Nutzung von Nanokristallen reduziert nicht nur die benötigte Menge der oft seltenen und teuren Proben, sondern erspart den Forschern zudem das Züchten großer Proteinkristalle, wie bei konventionellen Röntgenmethoden – oftmals eine extrem schwierige Aufgabe.
Um die vom Elektronenstrahl verursachten Schäden zu umgehen, wird statt eines Einzelbilds ein kurzer Film mit einer Hochgeschwindigkeitskamera gedreht, während der Elektronenstrahl auf einem Kristall ruht. Der Film zeigt, wie der Kristall buchstäblich dahinschmilzt. Aber dennoch bleiben ausreichende Informationen im „Diffraction during Destruction-
Außer bei Proteinen und anderen Biomolekülen kann die serielle Elektronenbeugung auch für viele neuartige Funktionsmaterialien eingesetzt werden – wie zum Beispiel Perowskite und metallorganische Frameworks, die vielversprechende Kandidaten für zukünftige Anwendungen in Solarzellen und Wasserstoffspeichern darstellen. Das Forschungsteam ist begeistert über die Einfachheit der Methode mit ihren verhältnismäßig geringen Anforderungen an wertvolle Proben und aufwändige Laborausstattung und erhofft sich ihre Verbreitung vom MPSD in Laboren weltweit.
MPSD / RK
Weitere Infos
- Originalveröffentlichung
R. Bücker et al.: Serial protein crystallography in an electron microscope, Nat. Commun. 11, 996 (2020); DOI: 10.1038/s41467-020-14793-0 - Abt. Dynamik in atomarer Auflösung, Max-Planck-Institut für Struktur und Dynamik der Materie, Hamburg
- Center for Free-Electron Laser Science, Deutsches Elektronen-Synchrotron, Hamburg











